学习报告(week 6)
SMPLify Demo Test 2
在上次运行 demo 的基础上,我们这次将尝试运行自己的测试样例,而不是 SMPLify 提供的 LSP dataset。
上次测试的时候,我们还留有一个问题尚未解决:在 SMPLify 上对 LSP dataset 进行测试后无法获得对应的 pkl 文件。在仔细研究了源代码后,我发现这个问题是 Python2 与 Python3 在读写文件上有差别所导致的。为了解决这个问题,我们需要先定位到 ‘fit_3d.py’ 中的:
with open(out_path, 'wb+') as outf:
pickle.dump(params, outf)
我们把这两行代码改成如下形式,即可获得保存关节点数据的 pkl 文件了:
with open(out_path, 'w+') as tmp:
tmp.write(params.__str__())
接下来,我们要尝试使用 SMPLify 测试我们自己的输入图片。
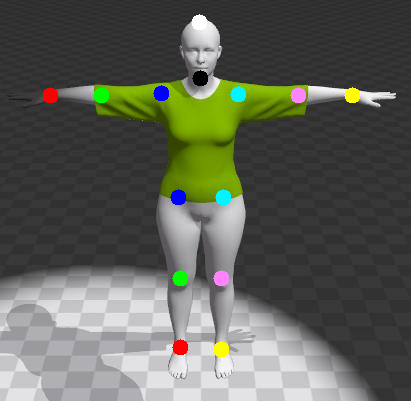
首先,我们需要获取输入图片的 DeepCut 预测关节点,经由 DeepCut 获得的 14 个关节点与 SMPLify 关节点的对应关系如下:
| index | joint name | corresponding SMPL joint ids |
|---|---|---|
| 0 | Right ankle | 8 |
| 1 | Right knee | 5 |
| 2 | Right hip | 2 |
| 3 | Left hip | 1 |
| 4 | Left knee | 4 |
| 5 | Left ankle | 7 |
| 6 | Right wrist | 21 |
| 7 | Right elbow | 19 |
| 8 | Right shoulder | 17 |
| 9 | Left shoulder | 16 |
| 10 | Left elbow | 18 |
| 11 | Left wrist | 20 |
| 12 | Neck | - |
| 13 | Head top | vertex 411 (see line 233:fit_3d.py) |
而 DeepCut 关节点的输出格式如下:
The pose in 5x14 layout. The first axis is along per-joint information,
the second the joints. Information is:
1. position x,
2. position y,
3. CNN confidence,
4. CNN offset vector x,
5. CNN offset vector y.
在此,我们需要用到的 DeepCut 关节点数据为其输出的前三维,即:x 坐标、y 坐标、预测 confidence。
在尝试安装 DeepCut 的依赖库 Caffe 时,我遇到了不少的问题,最终耗费了不少时间才成功运行 DeepCut。因此,我先简单描述一下 DeepCut 的环境配置操作。
最开始的时候,我分别尝试了在 macOS 与 Ubuntu 16.04 上安装 Caffe,具体教程可以参照以下网址。但是,Caffe 框架使用到了很多依赖库,我们简单地通过 pip3 安装这些依赖库的话,会出现各个依赖库间版本不兼容的问题,即在终端执行如下命令无法正确安装 Caffe 的依赖库:
$ pip install -r requirements.txt
为了解决这个问题,我尝试去安装一些过时了的版本的依赖库,希望能借此解决不兼容的问题。但是,有些库的资源已经不太好找到了,因此,我最后使用了一种操作上比较简单的方法解决了这个问题。我们需要先安装一个 Ubuntu 18.04 系统,然后在终端执行如下命令:
$ sudo apt install caffe-cpu
然后,我们从 DeepCut 的官方 Github 上下载它的源代码。我们根据 README 中的教程,在终端依次执行如下命令:
$ pip3 install click
$ cd models/deepercut
$ ./download_models.sh
而在 run demo 时,我们不能完全依照教程中的步骤来执行。由于我们安装的是 cpu 版本的 Caffe,我们要在终端执行如下命令以运行 DeepCut demo:
$ cd python/pose
$ python3 ./pose_demo.py image.png --out_name=prediction --use_cpu
DeepCut 的关节点将储存在 ‘prediction.npz’ 文件中。接下来,我们要运行 SMPLify 的 ‘fit_3d.py’ 来测试我们的输入图片。
首先,我们可以留意到 ‘fit_3d.py’ 中有如下代码:
with open(join(data_dir, 'lsp_gender.csv')) as f:
genders = f.readlines()
我们可以看到 SMPLify 需要读入一个保存测试图片性别的 csv 文件,因此,我们需要先为我们的测试图片创建一个这样的文件。
然后,SMPLify 需要读入 ‘est_joints.npz’ 文件,这个文件是一个 3x14xN 的 np 数组,其中,N 为测试图片的数目。在此,我们需要对如下代码进行一点修改:
est = np.load(join(data_dir, 'est_joints.npz'))['est_joints']
在 ‘prediction.npz’ 中,关节点被存放在 dict == ‘pose’ 中,我们要把代码修改成如下形式:
est = np.load(join(data_dir, 'prediction.npz'))['pose']
且外,由于我们的 prediction.npz 是 5x14 的 np 数组,我们还需要对其进行 reshape 才可以适用于 SMPLify 中:
est = est[0:3]
est = np.reshape(est, (-1, 14, 1))
至此,我们只需运行 ‘fit_3d.py’ 即可获得测试图片相对应的 SMPLify 预测结果:

显然,这个结果是存在明显的误差的,我们接下来需要定位误差产生的原因。
我们尝试使用 DeepCut 对 LSP dataset 的第一张图片进行预测:

然后将 DeepCut 的结果输入到 SMPLify(左图为自己运行 DeepCut 后得到的结果,右图为使用官方提供的 DeepCut 数据得到的结果):
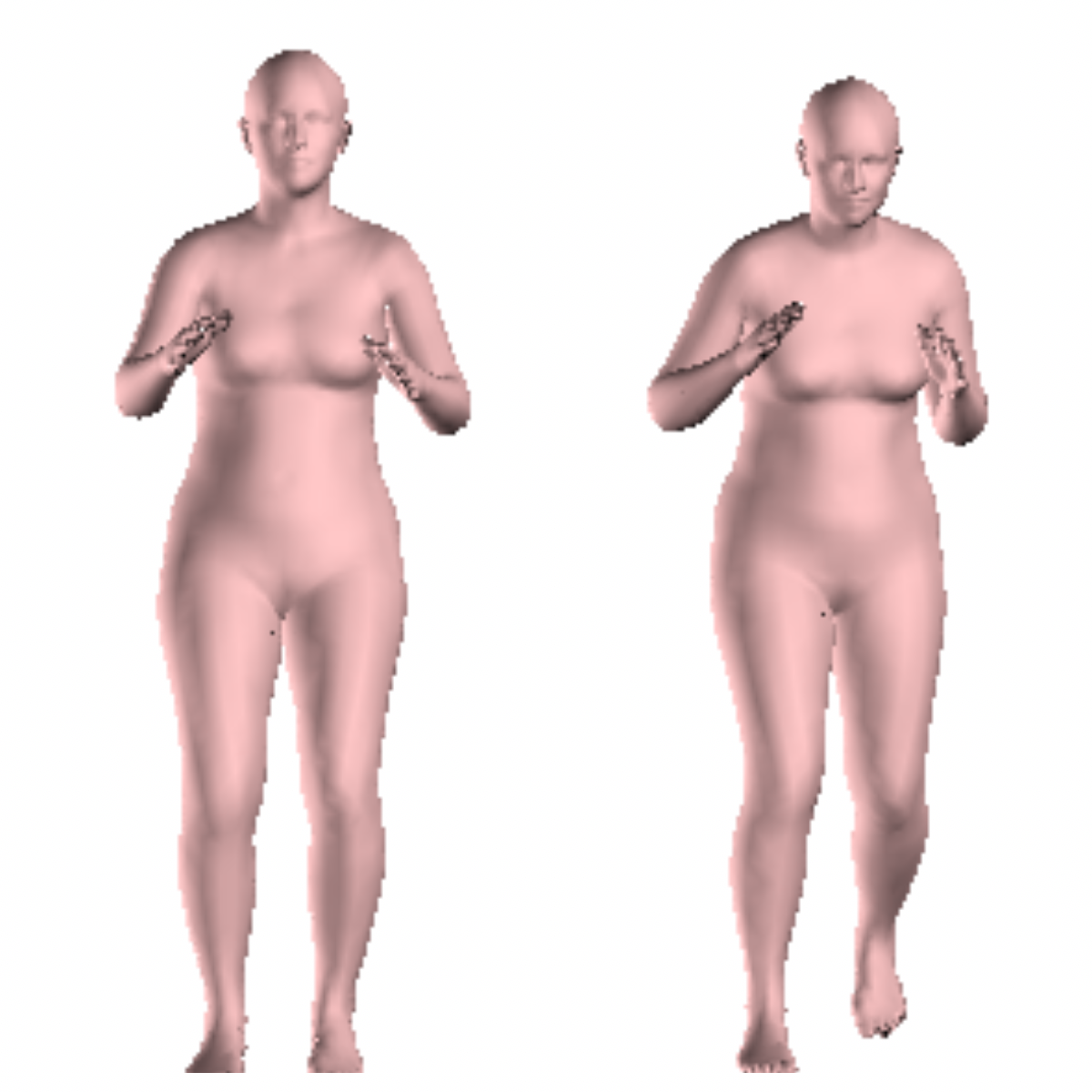
我们发现即便使用 LSP dataset,我们也无法获得与 SMPLify 官方一样的结果。因此,我将自己运行 DeepCut 的输出与官方提供的 DeepCut 输出分别打印出来(上图为自己运行的结果,下图为官方提供的结果):
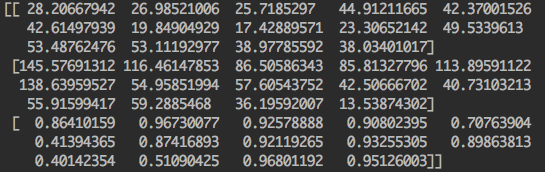
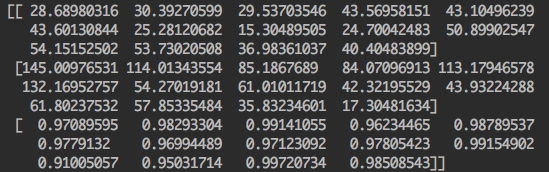
我们可以明显发现两者有较大的差别,因此,我们可以大胆推测目前阶段误差出现的原因是 DeepCut 的问题。
为了解决这个问题,需要先研究清楚 DeepCut 的论文,故目前还需要一点时间才能完成这个工作。我会在给课程期末项目收尾后,尽快完成这部分的工作。